|
|
|
|
|
深度学习在生物学中的应用 |
|
对MinION测序结果中base calling的计算 |
记得去年“阿尔法狗”(AlphaGo)的新闻出来后,有些生物学领域的研究人员可能会考虑是否需要跨专业学习一下Artificial Intelligence (AI),看看它能否在生物领域也掀起热浪。结果当他们刚刚了解到阿尔法狗的命脉乃来自Deep Learning (深度学习)真传时,它的亲兄弟“AlphaFold” 就以迅雷不及掩耳之势(2018年12月新闻发布会)在蛋白质折叠预测领域独领风骚。有生物学背景的人都知道,虽然科学家们破译了基因组,但从DNA到蛋白质翻译过程受各种基因和/或蛋白质的调控、修饰,并且蛋白质从翻译产生到能发挥功能的这一过程也是在细胞内经历了多次折叠。但人家“AlphaFold”则不畏这些千难万苦,“硬生生”的通过氨基酸序列直接预测蛋白质的3D结构(AlphaFold 的新闻发布链接:https://deepmind.com/blog/alphafold/)。所以当“AlphaFold”一出世,大家都惊呼它是能把诺贝尔奖抱回家的人选之一。
既然“AlphaFold”和“AlphaGo”是亲兄弟,那它们两个到底有什么相同点呢?其实这个相同点就是Deep Learing算法。而不同点则在于该算法分别应用在了围棋领域和蛋白质研究领域。所以,无论是生物,物理,还是化学领域的科研人员是时候学习一下Deep Learing算法,说不定你就是下一个诺贝尔获奖者呢!原来从事Deep Learing研究的专家和学者已经进军到生物领域的各个方向了。
Quantitative Biology (QB)期刊向来提倡交叉,尤其是计算、数学、物理前沿领域与生命科学的交叉,并且期刊也一直在跟踪刊登这些交叉领域的前沿热点文章。在Deep Learning这个火热阶段,QB编辑部邀请到了该领域的曾坚阳教授和裴剑锋教授作为QB期刊2018年第四期的Guest Editors,为QB刊组织了一场关于Deep Learning或Neural Network在第三代测序分析碱基识别、线粒体形态定量分析和药用蛋白预测中应用的“盛宴”(感兴趣的研究人员可以登陆期刊网站先睹为快:http://journal.hep.com.cn/qb/EN/2095-4689/current.shtml)。
今天重点推送的是Deep Learning在MinION测序仪base-calling中的应用【1】(WaveNano:a signal-level nanopore base-caller via simultaneous prediction of nucleotide labels and move labels through bi-directional WaveNets)。

文章简介:
这篇文章是来自沙特阿卜杜拉国王科技大学(KAUST)的Xin Gao教授团队与香港中文大学(深圳)的Zhen Li 博士合作完成。 Xin Gao 教授团队目前已经完成了一系列与纳米孔测序相关的工作。该团队关于Deep Learning在MinION测序仪数据模拟器中的工作(DeepSimulator: a deep simulator for Nanopore sequencing)于今年9月份发表在了生物信息学领域老牌期刊Bioinformatics上的哦【2】。此外,该团队还完成了一款全新的cwDTW算法,可以高效的联配超长的纳米孔信号,并以此为基础进行信号标注(signal labeling)从而能够检测单核苷酸多态性(SNP)【3】。该工作在国际顶级生物信息学会议ECCB 2018上口头展示,同时亦发表于Bioinformatics。在这里向Xin Gao教授及其团队表示祝贺!
英国生物技术公司Oxford Nanopore自2014年推出MinION测序仪后,由于其小巧的身材(iphone大小),要求不高的运行环境,较长的reads读取(超过15kb),较快的测序速度,实时的测序数据监测等特点,一经问世就受到广泛关注。该测序仪于2016年登上了国际太空站(ISS),完成了第一次太空测序,并证实了对Lambda phage的测序结果在ISS和地球上并无差别【4】。 MinION测序仪的基本工作原理是基于纳米孔测序技术,通过检测单链DNA分子通过纳米孔时引起电流变化的不同,用于碱基的识别(见Figure 1)。由于电流检测的频率通常是DNA序列通过纳米孔速度的7-9倍,因此这对base-calling造成巨大的技术挑战。此外,较高的测序错误率,尤其是对indels(插入和缺失)的测序,是纳米孔测序仪面临的一个主要问题。
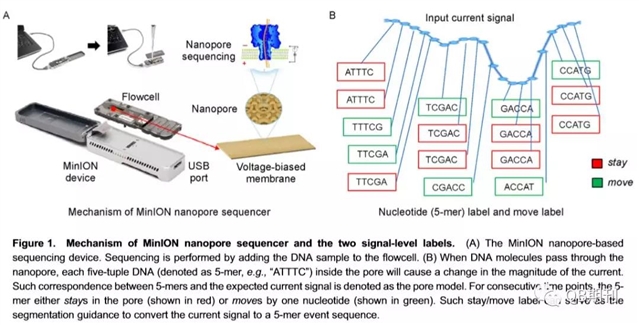
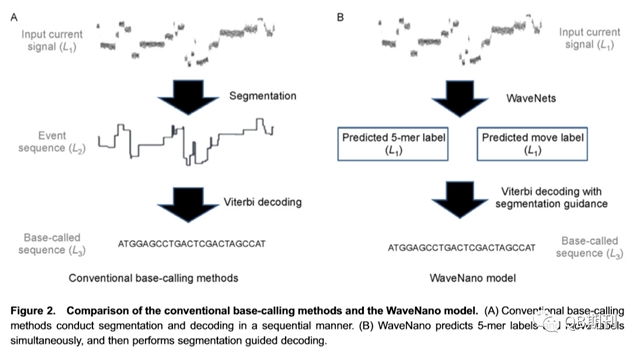
为了解决纳米孔测序仪较高错误率的问题,目前已经出现了多种算法。这些算法可以简单地归为两类,即基于机器学习(Machine Learning)的算法和基于共有序列(Consensus)的算法。这两种算法的基本原理都是通过serial base-calling过程(见Figure 2A)进行碱基识别,而这一过程势必会增加错误率。此外,这两种算法所用到的机器学习中的模型建构(Model Architecture)更适用于短片段的计算。为了解决上述问题,本文作者们采用了Google DeepMind团队在语音合成和语音识别方面新开发的具有完美表现的WaveNets深度学习方法【5】将纳米孔中的信号当作语音信号,而base-calling则类似于语音识别过程,开发了一种基于机器学习的新算法-WaveNano(见Figure 2B和Figure 3)。这种算法不依赖任何segmentations/decoding工具,而完全是一种self-contained 的线下工具。
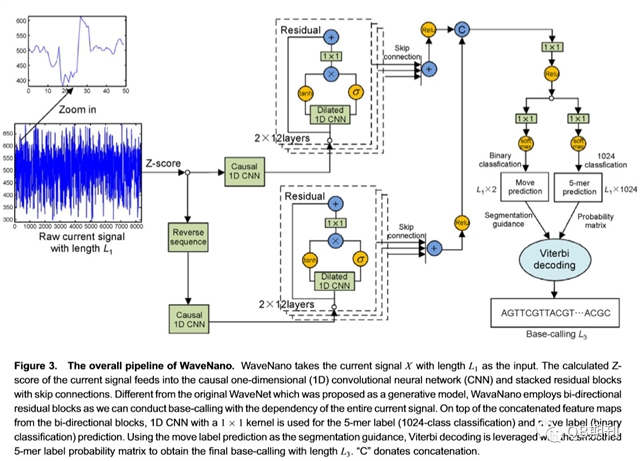
通过该算法,文章作者对Lambda phage的基因组用MinION进行了测序,结果得到了大约24,000个reads,电流信号平均为63,000bp。同时,作者还将WaveNano与官方的Metrichor算法以及Albacore算法进行了结果比较(如Table 1),结果表明WaveNano不仅能预测比较准确的DNA序列,同时该算法对indel的处理结果明显优于Metrichor和Albacore。此外,WaveNano的运行时间约为1个信号序列为0.5s,而Albacore的运行时间则为2s。
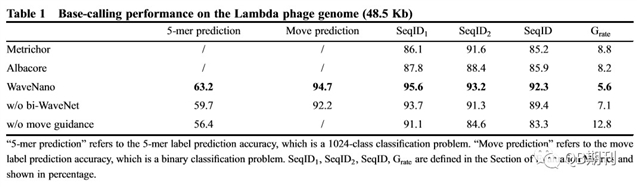
由此可见,WaveNano算法对于分析MinION产生的Lambda phage测序结果具有良好的表现,尤其对于indel序列的分析,其结果要比目前商用的Metrichor和Albacore具有更高的准确度。
Reference
1. Sheng Wang, Zhen Li, Yizhou Yu and Xin Gao. (2018) WaveNano:a signal-level nanopore base-caller via simultaneous prediction of nucleotide labels and move labels through bi-directional WaveNets. Quant. Biol., 6 (4): 359-368.
2. Yu Li, Renmin Han, Chongwei Bi, Mo Li, Sheng Wang, Xin Gao. (2018) DeepSimulator: a deep simulator for Nanopore sequencing. Bioinformatics, 34 (17), 2899-2908
3. Renmin Han, Yu Li, Xin Gao, Sheng Wang. (2018) An accurate and rapid continuous wavelet dynamic time warping algorithm for end-to-end mapping in ultra-long nanopore sequencing. Bioinformatics, 34 (17), i722-i731
4. Castro-Wallace, S. L., Chiu, C. Y., John, K. K., Stahl, S. E., Rubins, K.H.,McIntyre, A.B.R., Dworkin, J.P.,Lupisella, M.L., Smith, D. J., Botkin, D. J., et al. (2017) Nanopore DNA sequencing and genome assembly on the International Space Station. Sci. Rep., 7, 18022
5. Van Den Oord, A., Dieleman, S., Zen, H., Simonyan, K., Vinyals, O., Graves, A., Kalchbrenner, N., Senior, A., and Kavukcuoglu K. (2016) Wavenet: A generative model for raw audio. ArXiv, 1609.03499
关于期刊:
Quantitative Biology(QB)期刊于2013年创刊,由高等教育出版社、清华大学和北京大学共同主办。由教育部主管,高等教育出版社出版、德国施普林格(Springer)出版公司负责海外发行。
QB期刊主要报道生命科学的定量研究和系统整合的前沿热点,包括系统生物学、合成生物学、生物信息学以及计算生物学领域的最新研究成果和前沿进展,旨在推动定量生物学领域科研水平提升,同时为生命科学与计算机、数学、物理等交叉研究领域打造一个学术水平高、可读性强、具有全球影响力的交叉学科期刊品牌。
目前,QB以网络版和印刷版两种形式出版,全球免费下载。同时,被QB收录的文章全部免费出版,不收取任何费用。此外,对于特别优秀文章开通绿色通道,从投稿到第一个决定只需1周。
更多的文章请点击:
http://journal.hep.com.cn/qb
https://link.springer.com/journal/40484
欢迎投稿,投稿直通:
https://mc.manuscriptcentral.com/qb
主编阵容:
期刊目前由清华大学/美国德克萨斯大学达拉斯分校张奇伟教授和北京大学定量生物学中心汤超教授担任共同主编。清华大学张学工教授担任执行主编。此外,期刊还邀请了40余名国内外知名领域专家、学者组成编委会,保证刊物的高学术水平。

共同主编:张奇伟(Michael Q. Zhang)教授

共同主编:汤超 教授

执行主编: 张学工教授
(来源:科学网)
特别声明:本文转载仅仅是出于传播信息的需要,并不意味着代表本网站观点或证实其内容的真实性;如其他媒体、网站或个人从本网站转载使用,须保留本网站注明的“来源”,并自负版权等法律责任;作者如果不希望被转载或者联系转载稿费等事宜,请与我们接洽。