|
|
|
|
|
FDE 论文解读 | AI 驱动的基因组解码:大语言模型在基因组学中的应用 |
|
|
论文标题:AI-Empowered Genome Decoding: Applications of Large Language Models in Genomics
期刊:Frontiers of Digital Education
作者:Shaopeng Li, Weiliang Fan, Yu Zhou
发表时间:25 Mar 2025
DOI:10.1007/s44366-025-0051-1
微信链接:点击此处阅读微信文章

大语言模型(LLMs)在自然语言处理领域表现出色,而基因组学数据如DNA、RNA和蛋白质序列具有序列性质,与人类自然语言相似,这为LLMs在基因组学中的应用提供了基础。尽管LLMs应用于基因组学研究仅约四年,但已在许多具有挑战性的重要问题上取得了显著进展。
研 究 论 文
武汉大学周宇教授团队在Frontiers of Digital Education(《数字教育前沿(英文)》)期刊上发表了一篇题为AI-Empowered Genome Decoding: Applications of Large Language Models in Genomics的文章,围绕LLMs在基因组学中的应用展开综述,介绍了LLMs在基因组学研究中的应用进展、面临的挑战和发展前景。
基因组研究中使用的基础模型大多围绕蛋白质、DNA和RNA序列进行构建。蛋白质基础模型包括ProGen系列和ESM系列等模型,ProGen系列最初专注于蛋白质序列生成,在ProGen2中进一步增加参数规模,并针对抗体设计等常见任务进行了优化;ESM系列则在结构预测等任务上表现突出,与Alphafold等方法相比,ESMfold精度稍低,但速度更快。DNA基础模型包括Nucleotide Transformer和Evo等模型,Nucleotide Transformer通过掩码语言建模(MLM)学习基因组信息;而Evo利用状态空间模型(SSM)获得更长的模型上下文长度,以更有效地发现长序列中的模式。RNA基础模型中,RNAErnie使用ERNIE的多阶段掩码策略和基于类型的微调提升对RNA序列的理解能力,而DGRNA等模型则通过更大的数据集和更新的模型架构提升预测性能。此外,还有针对特定研究领域的基础模型,如UTR-LM和SpliceBERT。
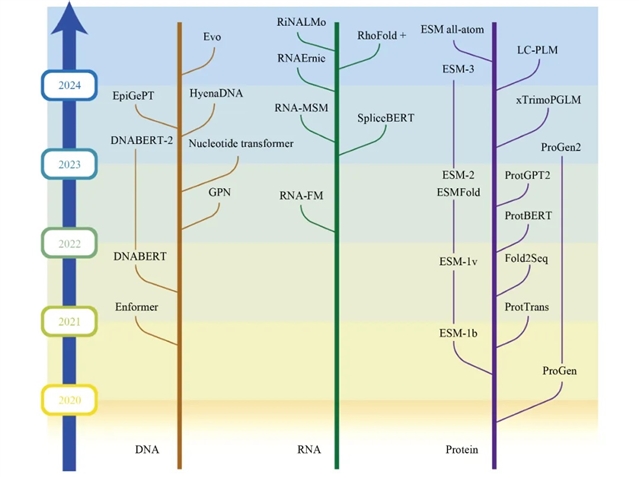
图1 基因组学中DNA、RNA及蛋白质语言主要大语言模型发展简史
大语言模型同样广泛应用于基因组分析的其他任务,BioLLMNet可用于RNA-RNA和蛋白质-RNA相互作用预测,其利用多模态方法结合不同生物分子的语言模型编码;scBERT、GPTCelltype等模型可用于单细胞数据分析中的细胞类型注释等任务;scFoundation进一步在药物反应预测、Perturb-seq结果预测等其他单细胞数据下游分析任务中达到先进水平;在生物分子结构预测方面,LLMs在蛋白质和RNA结构预测中均有应用,ESMFold在蛋白质结构预测中兼具速度和准确性,RNA-MSM、RNAformer等在RNA结构预测中也取得了进展。
文章还讨论了LLMs在基因组学应用中面临的挑战与发展前景,目前主要的挑战包括生物数据与人类语言的本质差异导致自然语言模型的先进技术难以直接应用,以及模型规模增长带来的计算资源需求问题;而近期的发展点在于生物数据编码和特定提示工程方法的改进,以及面向更多下游任务的多样化模型开发。此外,为帮助学生了解与应用生物大语言模型,推进大模型在基因组学研究中的应用,文章提出了将LLMs整合到基因组学课程中的八个实践项目,涵盖序列生成、基因注释等多个方面。总之,随着LLMs的不断发展,其在基因组学中的应用将进一步拓展,为基因组学研究带来新的机遇和突破。
文 章 信 息
Shaopeng Li, Weiliang Fan, Yu Zhou. AI-Empowered Genome Decoding: Applications of Large Language Models in Genomics. Frontiers of Digital Education, 2025, 2(1): 14
https://doi.org/10.1007/s44366-025-0051-1

识别二维码,免费获取原文
作 者 信 息

李少鹏,博士后。主要关注于神经网络在生物信息处理中的应用,侧重于基于神经网络的生物图像处理、多组学分析等方面,相关成果以第一(含共同)作者发表于相关领域的知名学术期刊。

樊伟良,博士研究生。主要关注语言模型与图神经网络在生物数据处理中的应用,以及生物信息数据的可视化工具;以第一(含共同)作者在知名学术期刊发表多篇论文。

周宇,教授、博士生导师。长期从事生物信息与RNA生物学研究,近年来重点围绕RNA加工的调控与功能开展研究,发现了RNA渐进式加尾这一新模式及其调控转录后m6A修饰的功能,揭示了RNA加工和m6A修饰分别调控细胞核和细胞质RNA分选的新机制,解析了RNA加工与调控在精子发生、细胞应激等生理病理过程的重要功能。以通讯(含共同)作者在知名学术期刊发表研究论文20余篇,其中多篇获亮点专评、F1000推荐;获批基金委优青等项目支持。
往 期 回 顾
论文解读 | 王学男:DeepSeek的中国式创新重塑中国教育自信
论文解读 | 基于知识图谱的专业课程的教学设计与实践
论文解读 | 大学生人工智能素养评价体系的构建
论文解读 | 学习的演变:评估 GenAI 对高等教育变革的影响
前沿观点 | 张立国:人工智能教育伦理问题及其规约
期 刊 介 绍
期刊特点
1. 国际化投审稿平台Editorial Manager方便快捷。
2. 严格的同行评议(Peer Review)。
3. 免费语言润色,有力保障出版质量。
4. 不收取作者任何费用。
5. 不限文章长度。
6. 审稿周期:第一轮平均30天,投稿到录用平均60天。
7. 在线优先出版(CAP)。
8. 通过Springer Link平台面向全球推广。
在线浏览
https://journal.hep.com.cn/fde
(中国大陆免费下载)
https://link.springer.com/journal/44366
在线投稿
https://www.editorialmanager.com/fode/
邮发代号
80-164
联系我们
fde@hep.com.cn
010-58582344, 010-58581581


特别声明:本文转载仅仅是出于传播信息的需要,并不意味着代表本网站观点或证实其内容的真实性;如其他媒体、网站或个人从本网站转载使用,须保留本网站注明的“来源”,并自负版权等法律责任;作者如果不希望被转载或者联系转载稿费等事宜,请与我们接洽。