随着生物测序技术突破,全球天然基因库已积累数十亿量级序列,其中蕴藏海量高价值功能基因。然而,当前仅有少数明星基因被深度挖掘,绝大多数仍处于“沉睡”状态。如何突破传统注释与建模局限,利用AI等手段激活“基因宝库”,正成为合成生物学与生物制造领域的关键挑战。
4月9日,中国科学院深圳先进技术研究院定量合成生物学全国重点实验室、合成生物学研究所娄春波团队与北京大学定量生物学中心钱珑团队合作在国际学术期刊Science Advances上发表题为"Discovery of Diverse and High-quality mRNA Capping Enzymes through a Language Model-enabled Platform"的研究论文,报道了全球首个面向合成生物学元件挖掘与生物制造应用的大语言模型——"SYMPLEX",并将SYMPLEX模型应用于mRNA加帽酶基因的挖掘,展示了大语言模型赋能生物制造的巨大潜力。
该模型通过融合领域大语言模型训练、合成生物专家知识对齐和大规模生物信息分析,实现了从海量文献中自动化挖掘功能基因元件,并精准评估其工程化应用潜力。研究团队将SYMPLEX应用于mRNA疫苗生物制造关键酶——加帽酶的挖掘,成功获得多种高性能新型加帽酶。第三方公司实验验证显示,这些酶在催化效率上超越国际头部企业New England Biolabs(NEB)商业化加帽酶2倍以上,显著提升了mRNA疫苗生产率和成本效益。此项成果不仅为合成生物学元件设计提供了AI驱动的新范式,更展现了大语言模型等人工智能技术在生物制造中的广阔应用前景。
■破局传统:功能基因深度挖掘的大语言模型
天然生物基因组蕴藏着海量功能基因,这些基因在进化过程中不断优化,形成了多样化的序列空间和复杂精巧的功能活性,赋予生物体适应复杂环境的独特优势。随着高通量测序技术的发展,全球生物序列数据库已突破数十亿规模,为生物制造和合成生物学提供了前所未有的基因元件资源库。然而,尽管这些天然基因蕴含着巨大的应用潜力,目前仅有少数明星基因(如基因编辑工具酶)得到了系统的注释和结构解析。这种研究的不均衡导致现有基于序列、结构或深度学习的基因挖掘技术和蛋白质设计方法难以应用于更复杂的基因系统,严重制约了高价值功能基因的开发与利用。
针对上述问题,研究团队创造性地将大型语言模型(LLM)与结构化生物知识库深度融合,开发出SYMPLEX智能基因挖掘平台(图1)。SYMPLEX是强大的功能基因搜索引擎,通过自动化阅读和理解千万级体量的生物学文献,在基因、功能和知识水平上提取分析文献内容,并与专家数据库进行概念对齐、交互和基于先进生物信息技术的统计模式生成,从而提供证据链完整的高质量候选基因集合。SYMPLEX不仅有效规避了大语言模型幻觉,还能自动生成基因功能相关的细粒度知识树,引导科学家探索广泛的生物机制和分子过程(图1)。
对比结果表明,SYMPLEX大模型在挖掘基因的深度、数量和多样性上均显著优于传统生物信息学方法,其挖掘的基因多样性也超越了现有蛋白质功能预测模型的边界(图2)。
■应用案例:解锁mRNA疫苗高效生产的蛋白质密码
近年来,mRNA疫苗以其高效、可快速开发等特点在全球抗疫中发挥了关键作用。然而,mRNA疫苗背后的一项关键工艺——mRNA 5’端加帽(capping),却因其效率较低、成本高昂成为“卡脖子”环节。加帽过程对稳定mRNA、促进翻译和减少免疫反应至关重要,而目前mRNA疫苗生产工艺中使用的仍是传统的痘病毒双蛋白(Vaccinia D1/D12)加帽酶,选择有限且价格昂贵。
研究团队利用SYMPLEX大规模挖掘mRNA加帽酶,并进行了实验验证。SYMPLEX通过批量处理生物学文献和生物信息分析,识别出16,685个与 mRNA 加帽相关的基因,并进一步筛选出75类(18,779 条序列)高置信度的完整加帽酶基因。经过46种候选基因实测,研究团队获得了14种可在哺乳动物和酵母细胞中稳定发挥作用的加帽酶,其中2种新型加帽酶的体外活性比商业化痘病毒加帽酶高出两倍(图3)。值得一提的是,本研究挖掘的新型加帽酶与已知加帽酶的序列相似性低于20%,且编码序列长度缩减30%,有望为mRNA疫苗和基于mRNA的基因疗法研究提供关键使能技术支持。
此外,SYMPLEX的挖掘还揭示了加帽酶在自然界中多样的构型与进化策略。研究发现,加帽酶TPase功能域的桶状外围结构具有保守与可设计区间,而MTase功能域则存在新的酶活中心模体。这些发现表明,SYMPLEX不仅能助力于理解生物过程的多样化策略,还可为酶的理性工程优化或生成式蛋白设计提供高质量数据集。
■平台赋能:合成生物制造的“智能基座”
目前,SYMPLEX在线交互式平台已上线供研究人员免费使用(https://bdainformatics.org/page?type=SYMPLEX)(图4)。平台采用模块化设计,提供三个核心功能:
(1)文献智能提取引擎PubEngine:支持高通量的文献智能检索分析与可视化交互;
(2)基因功能标注系统GeneTagger:实现从分子机制到生物过程的细粒度自动化基因与功能提取;
(3)标准化知识中枢GeneNorm:实现与专家知识库的概念对齐与标准化,支持知识树构建和功能模式识别。
各模块既可无缝协同实现高效数据流转,又能独立运行,以加速功能基因挖掘以及蛋白质设计。平台现有注册用户200余人,2024年访问量达6000余次。
本项研究开创了功能基因深度挖掘的新范式,利用大语言模型高效推动生物知识转化,为mRNA疫苗规模化生产提供了关键酶资源库。研究团队正利用SYMPLEX挖掘更多可用于生物制造和合成生物学的关键酶元件,并将该平台拓展至合成通路设计等领域,有望推动生物制造进入“AI for Science”新纪元。
北京大学研究员钱珑、中国科学院深圳先进技术研究院研究员娄春波为本文共同通讯作者。北京大学博士研究生王天泽、覃博文、厉思宏,中国科学院深圳先进技术研究院博士研究生王子陌为共同第一作者。本研究获得了浙江大学欧阳颀教授团队和北京远轩科技有限公司的大力支持,并得到国家重点研发计划、国家自然科学基金、北京市重点基金以及深圳合成生物学创新研究院等项目的资助。
?
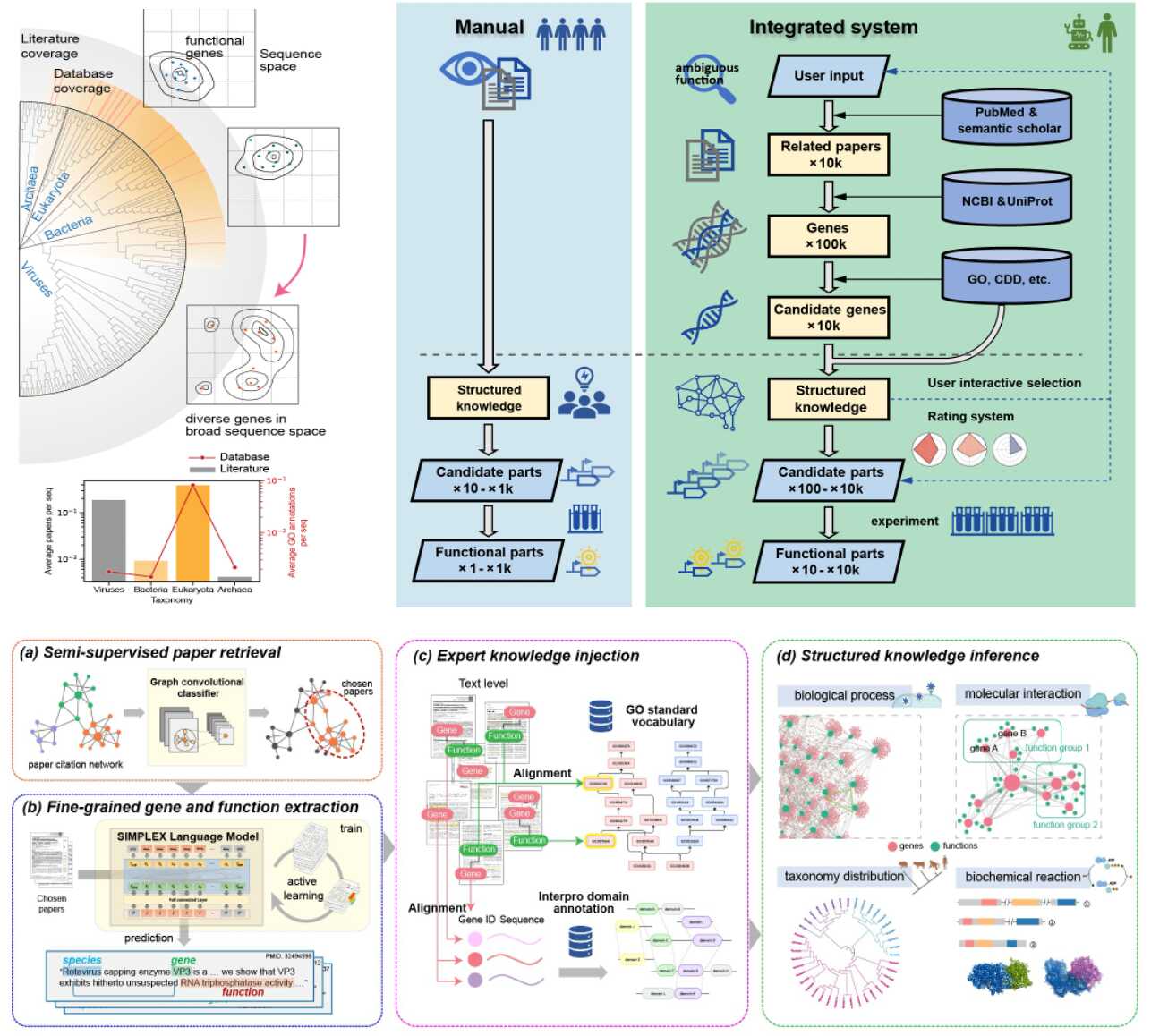 图1.SYMPLEX大模型的技术路线及其与传统基因挖掘流程对比
图1.SYMPLEX大模型的技术路线及其与传统基因挖掘流程对比
?
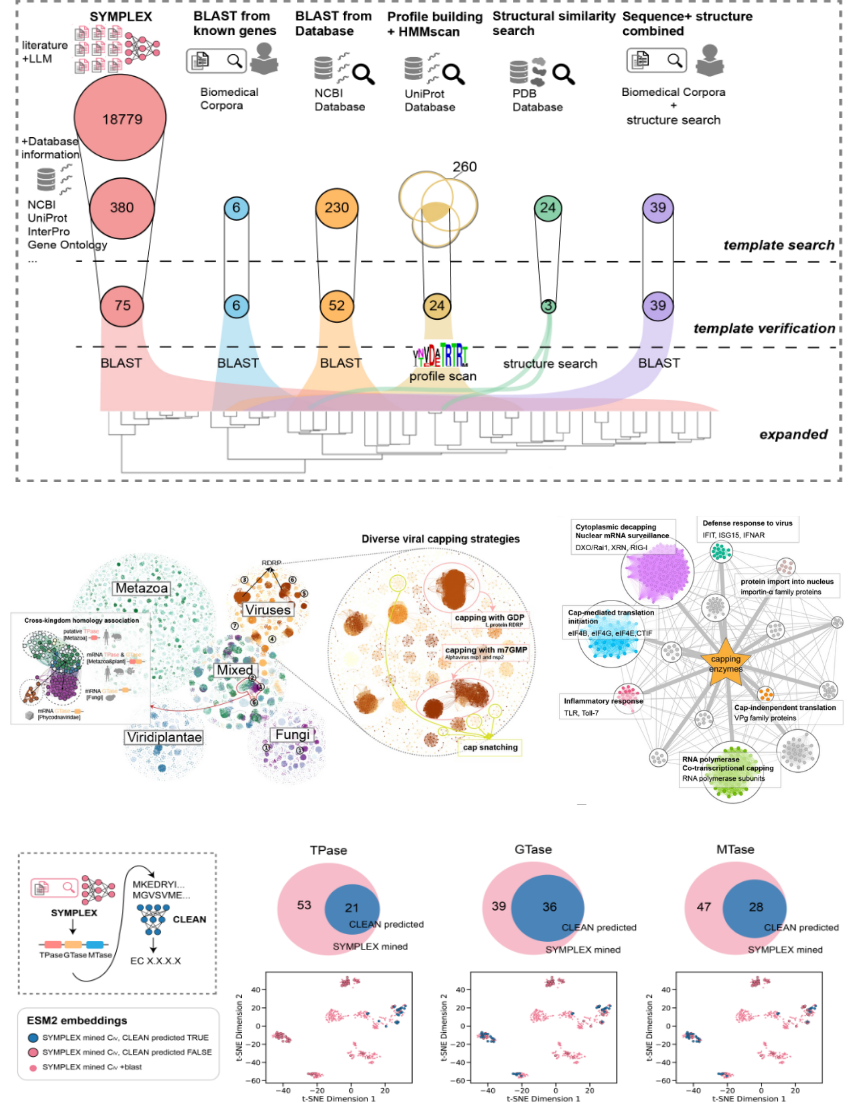 图2.SYMPLEX挖掘结果多样性对比和细粒度知识树生成
图2.SYMPLEX挖掘结果多样性对比和细粒度知识树生成
?
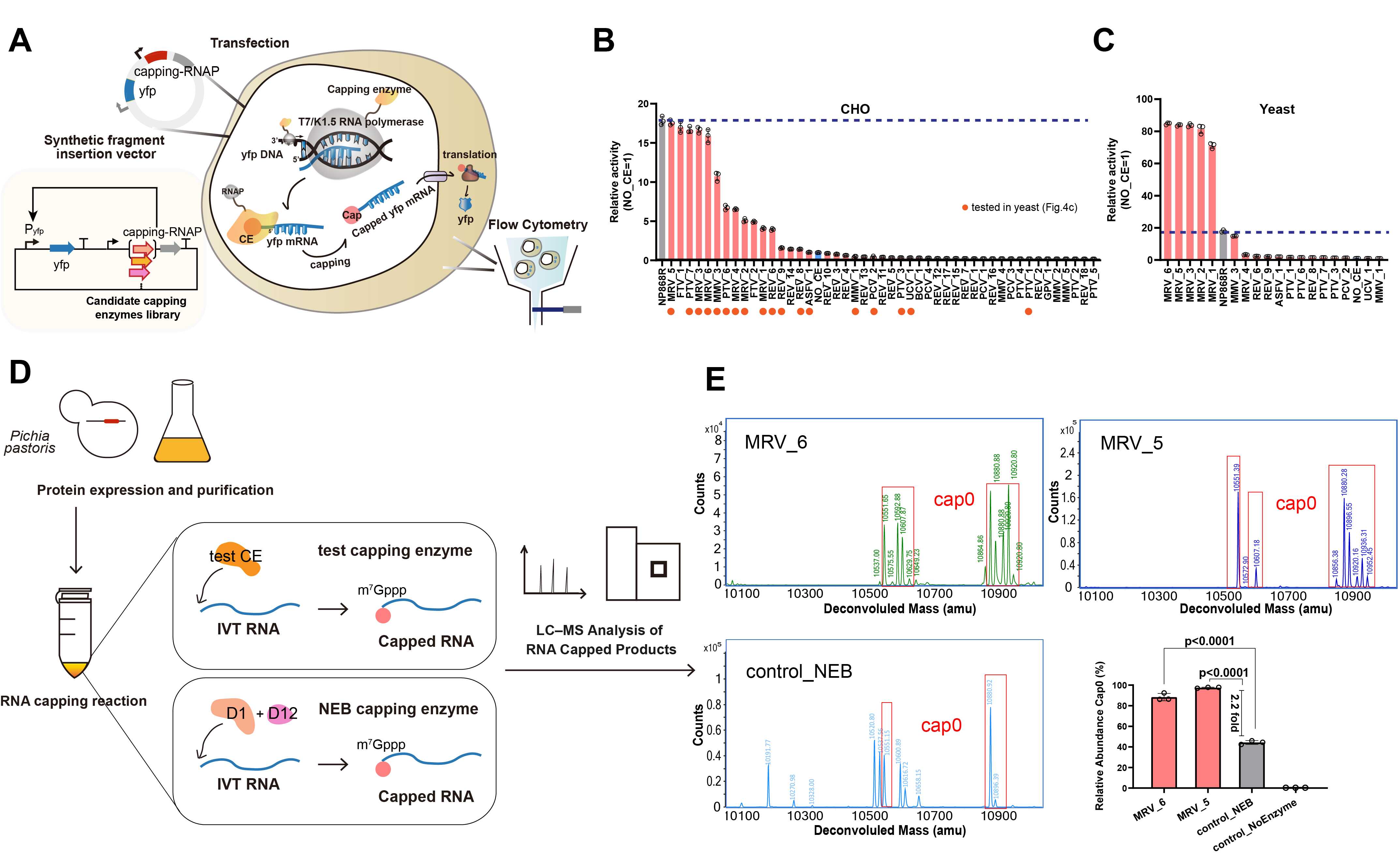 图3.候选加帽酶在细胞体系和体外转录体系中表现出跨物种、跨体系的高加帽效率
图3.候选加帽酶在细胞体系和体外转录体系中表现出跨物种、跨体系的高加帽效率
?
 图4.SYMPLEX平台
图4.SYMPLEX平台
(原标题:Science Advances | 功能基因智能挖掘大模型SYMPLEX推动生物制造与合成生物元件开发)
特别声明:本文转载仅仅是出于传播信息的需要,并不意味着代表本网站观点或证实其内容的真实性;如其他媒体、网站或个人从本网站转载使用,须保留本网站注明的“来源”,并自负版权等法律责任;作者如果不希望被转载或者联系转载稿费等事宜,请与我们接洽。