近日,西北工业大学计算机学院教授尚学群团队在单细胞与空间转录组数据整合分析方面取得新研究进展,研究成果发表于《创新》。
在生命医学研究中,精确解析细胞在组织中的空间分布与功能联系对于理解器官结构及疾病演进至关重要。单细胞转录组学测序技术虽能描绘单个细胞的基因表达全貌,但导致原始空间位置信息丢失;而空间转录组学测序技术能够保留细胞的空间信息,却面临测序分辨率低或检测基因数量受限的固有技术限制。如何有效整合两者的技术优势,克服现有测序数据的局限性,是当前计算生物学亟待解决的难题。
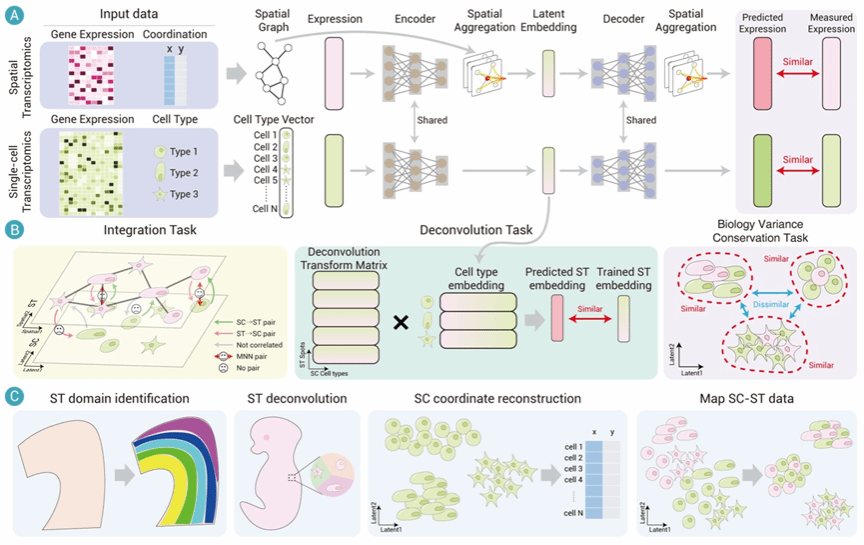 深度神经网络框架方法总体框架。西北工业大学供图
深度神经网络框架方法总体框架。西北工业大学供图
为了解决这些挑战,尚学群团队提出了基于多任务优化策略的深度神经网络框架(stSCI)。该方法通过设计异构数据融合模块,利用共享图注意力网络作为编码器,将多源异构的SC和ST数据投射到统一的、经批次校正的低维嵌入空间中。通过这种特征融合机制,模型在有效消除异构数据间技术差异与噪声干扰的同时,最大程度地保留了细胞间真实的生物学差异与拓扑结构特征。
实验结果表明,模型在空间域识别、三维组织结构解析、细胞类型反卷积及单细胞空间位置重构等多个关键任务中,综合性能均优于现有主流方法。通过将该方法应用于沙门氏菌感染的小鼠结肠模型测序数据,研究团队不仅成功识别出关键的淋巴组织区域,还精准捕捉到该区域在感染过程中表现出的“延迟激活”动态响应模式,这一发现与已知的感染免疫动力学高度吻合。该工作为解析复杂组织微环境、理解疾病演进机制等生物学研究提供了高效的人工智能方法模型。
相关论文信息:https://doi.org/10.1016/j.xinn.2025.101220
版权声明:凡本网注明“来源:中国科学报、科学网、科学新闻杂志”的所有作品,网站转载,请在正文上方注明来源和作者,且不得对内容作实质性改动;微信公众号、头条号等新媒体平台,转载请联系授权。邮箱:shouquan@stimes.cn。