在国家重点研发计划等项目资助下,我国科学家成功构建出全球首个整合五大组学的综合可视化兰科数据库——OrchidMD。这一成果为兰科植物的遗传机制解析、功能基因挖掘以及分子设计育种提供了极为关键的数据支撑与分析平台。11月11日,相关成果在线发表于《植物生物技术杂志》。
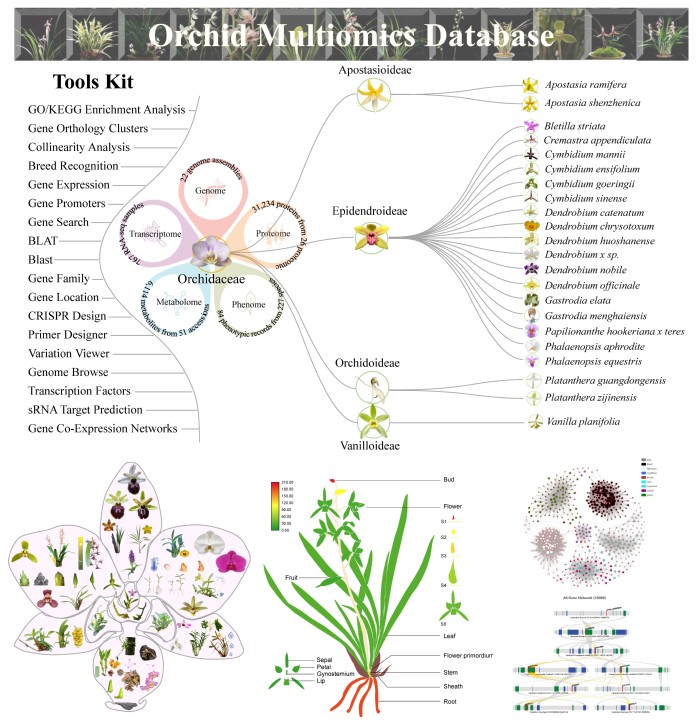
OrchidMD数据库的系统框架与部分功能示意图。研究团队供图
据介绍,OrchidMD数据库(访问地址:http://www.orchidcomics.com)实现了基因组、转录组、蛋白组、代谢组和表型组五大组学数据的深度整合。其数据覆盖范围广泛,涵盖了213种兰科植物,总数据量高达329.4 GB。该平台精心设置了23个功能模块,涵盖基因浏览、表达分析、共表达网络构建、GWAS关联分析、CRISPR靶点设计、基因家族分析等多个方面,能够为科研工作者提供一站式、全方位的多组学数据查询与可视化分析服务,极大地提升了科研效率。
该数据库在数据资源方面极为丰富且优质。它汇集了22个高质量的兰科植物基因组、767个转录组样本、26个蛋白质组数据集以及51个代谢组数据集。同时,还整合了69,224,698个遗传变异位点以及84个关键表型性状数据。如此庞大且高质量的数据资源,为兰科植物的分子标记开发、功能基因挖掘以及育种研究筑牢了坚实的根基。
针对兰科植物种类繁多、形态复杂等难题,广东省农业科学院环境园艺研究所研究员杨凤玺/朱根发团队、广东省农业科学院水稻研究所研究员刘琦团队以及大理大学教授杨邓奇团队强强联合,基于卷积神经网络与特征增强算法,成功开发出品种识别模块。该模块实现了兰属植物的自动识别,识别准确率高达86.5%。用户只需上传兰科植物图像,系统便能迅速识别并返回最相近的品种信息,显著提升了品种鉴定效率,为兰科植物的分类与研究提供了有力工具。
为充分验证OrchidMD数据库的实际应用价值,研究团队以ARF转录因子家族为研究对象展开深入探索。研究发现,CsiARF04在花芽及根发育过程中呈现高表达状态。通过转基因实验进一步证实,CsiARF04的过表达能够显著促进根状茎分化与新芽萌发,这一发现为解析兰科植物生长发育调控机制提供了全新的依据。
此外,该数据库集成了专为兰科植物优化的CRISPR基因编辑靶点设计工具。以墨兰为例,研究团队成功预测并验证了CsiPDS基因的编辑靶点,实现了我国传统国兰的精准编辑,填补了该领域的研究空白,为后续的功能验证与分子育种开辟了崭新的路径。
相关论文信息:https://doi.org/10.1111/pbi.70445
版权声明:凡本网注明“来源:中国科学报、科学网、科学新闻杂志”的所有作品,网站转载,请在正文上方注明来源和作者,且不得对内容作实质性改动;微信公众号、头条号等新媒体平台,转载请联系授权。邮箱:shouquan@stimes.cn。